ExPASy資料庫簡介-蛋白質序列分析與預測(2)
口述:呂平江 老師
整理:林崇文、鄭詩思、李佩真
Translate

![]()
3.選擇Translate
![]()

4.貼上欲查詢之基因序列
5.選擇TRANSLATE SEQOENCE
![]()
![]()
6.得到六個Frame
7.選擇其中一個作為蛋白質氨基酸之序列(通常挑Stop codon愈少者)
![]()

8.選擇5’3’Frame2之結果

BLAST
1. 連結到http://tw.expasy.org/tools
2. 在Similarity searches中選擇BLAST

![]()

Q D A V A S K I L G L P T Q T V D S S Q G S E Y D Y V I F T Q T T E T A H S C N V N R F N V A I T R A K I G I L C I M S D R D L Y D K L Q F T S L E I P R R N V A T L Q A E N V T G L F K D C S K I I T G L H P T Q A P T H L S V D I K F K T E G L C V D I P G I P K D M T Y R R L I S M M G F K M N Y Q V N G Y P N M F I T R E E A I R H V R A W I G F D V E G C H A T R D A V G T N L P L Q L G F S T G V N L V A V P T G Y V D T E N N L
3. 貼上PROTEIN sequence
4. 按Run BLAST
![]()
![]()
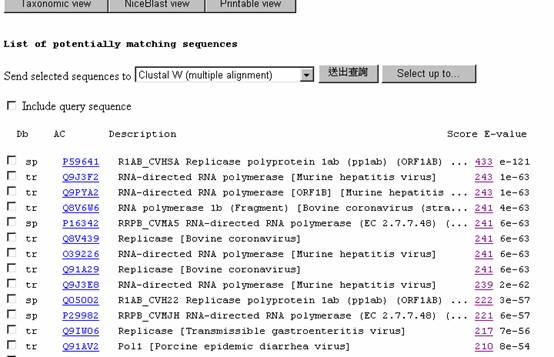
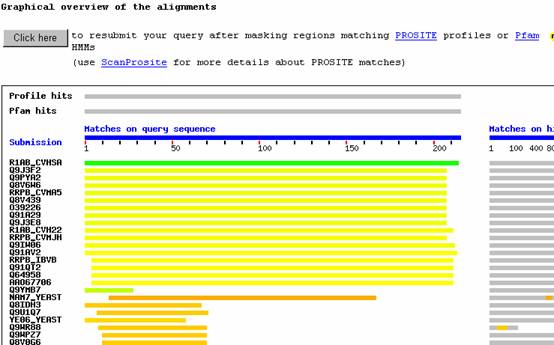
5. 比對成果如下
6. 依據Score及E-value之結果,選擇其一繼續比對,此以P59641為例
(通常是Score愈高而E-value愈低)
![]()
![]()
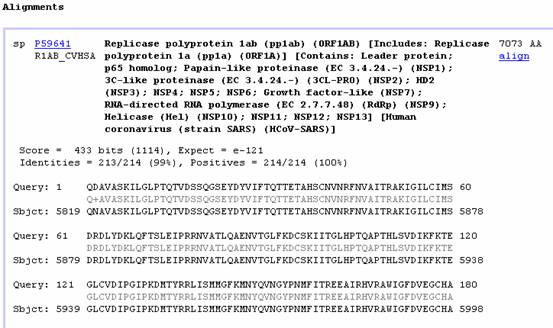
7. 以下為P59641之查詢結果
Primary structure(蛋白質的基本特性)
(一)ProtParam
1. 首先連結到http://tw.expasy.org/tools
2. 在Primary structure選擇ProtParam
![]()

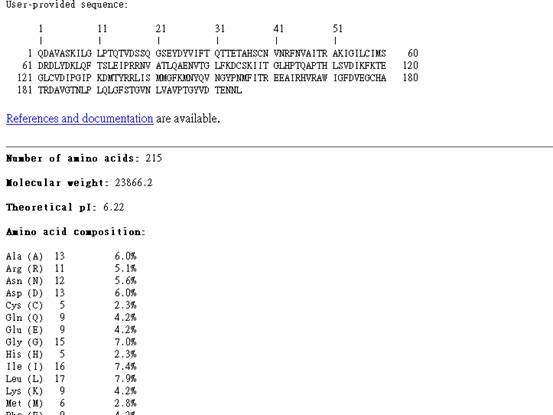
3. 貼上欲查詢之氨基酸序列之後按下Computer parameters
![]()

![]()

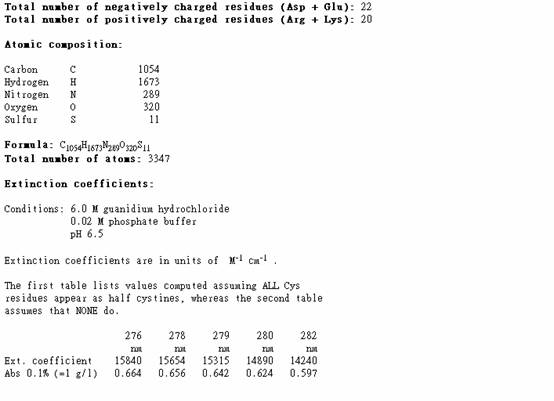
4.可得到此序列之pI值及Mw等資料

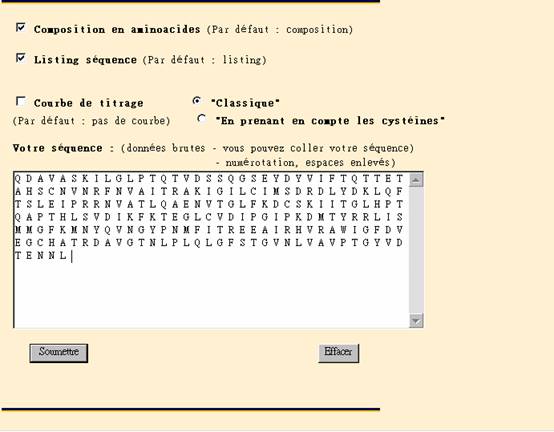
(二)MW,pI,Titration curve
1. 在Primary structure選擇MW,pI,Titration curve

![]()

2. 貼上欲查詢之氨基酸序列之後按下Sournettre




(三)ProtScale
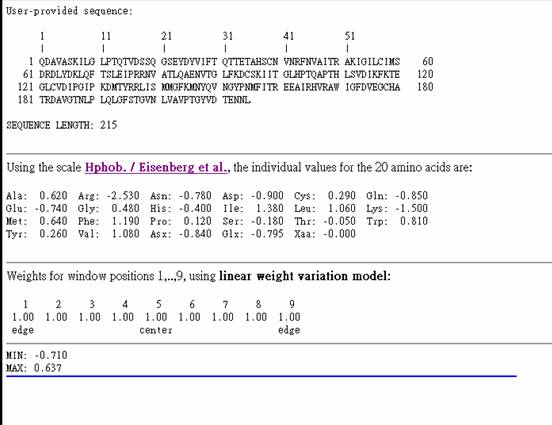
1. 在Primary structure選擇ProtScale
![]()
2. 貼上欲查詢之氨基酸序列
3.選擇amino acid scale
4.按下Submit
![]()


![]()
![]()

![]()
![]()


5.結果如下
![]()
![]()
![]()
![]()

TagIdent
1. 首先連結到http://tw.expasy.org/tools
2. 在Protein identification and characterization選擇TagIdent
![]()

3. 在下工具填上pI & Mw
4. 並按下Start TagIdent
![]()
![]()
![]()

![]()
![]()
![]()
![]()
![]()

![]()
![]()
![]()
![]()

5. 在Database Swiss-Prot中查詢到兩筆資料
6. 選擇VNS3_CVPFS繼續查詢
![]()


7.結果如下

補充說明(以window size為例)
1. 首先連結到http://tw.expasy.org/tools
2. 選擇Search ExPASy


3.填上window size按Enter開始查詢
![]()
![]()
![]()

4.查詢結果中選擇第一個(用紅色圈起處)


5.將可看到Window size及Weight的補充說明



