Cell Surface Display
整理:王永順
在篩選蛋白質的方法之中,還可以利用細胞表面表現(cell surface display),這是將所欲表現的蛋白質,表現於活的細胞膜外面,再利用fluorscence-activated cell sorting(FACS)篩選出表面帶有有所要的蛋白質的細胞,又因FACS有較低的背景值,能夠作很精細的篩選。而運用此種方法必須要將重組蛋白(recombinant proteins)基因接於寄主(host)細胞表面鑲嵌蛋白(anchor protein)基因上,利用寄主的系統將蛋白質轉譯出來並且表現於寄主細胞膜外面,再利用受質(substrate)篩選出正確表現的寄主細胞。到目前為止,在原核及真核細胞中,都找到了細胞表面鑲嵌蛋白的基因,而且被廣泛的應用 [1]。雖然以噬菌體(bacteriophage),表現重組蛋白於其外鞘蛋白(coat protein)早就已經建立且運用了,但是因受限於噬菌體的大小,所插入的(insert) DNA大小有所限制,無法利用其表現分子量較大的蛋白質;但是藉由原核或真核細胞表面表現,則可以表現分子量較大的蛋白質,而且也具蛋白質修飾作用,使得所表現出的蛋白質更符合需求 [4]。
在細菌(bacteria)中,主要是將重組蛋白質(recombinant proteins)表現於革蘭氏陰性細菌(Gram-negative bacteria)的細胞表面,其需要下列幾個特性:(1)能夠正常的被細胞製造並分泌到細胞質(cytoplasme)。(2)能夠找到細胞的外膜(outer membrane)。(3)能夠穿透外膜並鑲嵌於細胞表面。而在大腸桿菌(Escherichia coli)已經找到LamB、OmpA、OmpC、OmpS、OmpT、FhuA及the lipoprotein TraT等外膜蛋白;在1992年,Francisco等人,成功地將lipoprotein(Lpp)的outer membrane localization domain接於outer membrane protein A (OmpA)的46-159胺基酸,生成Lpp-OmpA hybrid (此即為anchor protein),並在其C端接上recombinant polypeptide,讓其表現於細胞表面;此為Lpp-OmpA system,也是第一個成功將全長的異種蛋白(full-length heterologous proteins)表現於大腸桿菌的表面。而這套系統接著被廣泛的利用,例如:cellulases、esterase、organophosphate hydrolases、scFv enzymes、-lactamase、thioredoxin等等都成功的表現出來 [1,2]。
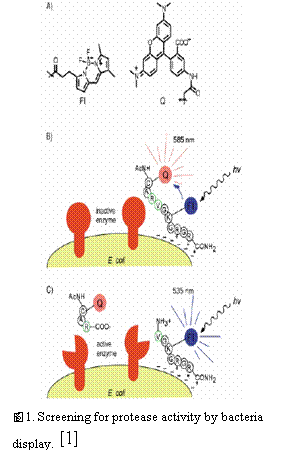
最近,利用細菌細胞表面表現(bacterial cell surface display)已經被用來篩選具活性的酵素(enzymatic activity)。隨著更多的研究,在2000年,Olsen等人利用E. coli OmpT protein library篩選具有有活性的酵素。OmpT為integral outer membrane protease,其大小為33.5 kDa,是一種serine protease,也與致病性有關,特別喜歡切Arginine(Arg, R)及Lysine(Lys, K)的位置。但在這裡,Olsen等人想找突變的OmpT,其只切Arg及Valine(Val, V)的位置。受質給予一段peptide,在其上面再加上fluorophore(Fl)及quenching fluorophore(Q),如圖 SHAPE \* MERGEFORMAT 1之A所示,這兩者作為分子內fluorescence resonance energy transfer(FRET)。受質設計為在Fl那邊帶+3電荷,受質與大腸桿菌共同培養時,當大腸桿菌表面呈現負電荷時,則受質就會因為正負電相互吸引而被吸附於細胞表面;假若細胞表面為不具活性的酵素(inactive enzyme),則對於受質上Arg及Val的切位不會作用,此時以 hv照射,Q能將Fl的螢光吸收,在發出波長585nm;如果細胞表面為具活性的酵素(active enzyme),則會對受質上Arg及Val的切位作用,使的帶有Q的peptide從受質上被切除,而帶有Fl的peptide因帶正電還被吸附於細胞表面,以 hv照射,Fl會發出波長535nm;再利用FACS分離並收集發出535nm的細胞。Olsen等人利用此方法從1.9×106的細胞中篩選出352個已經成功轉殖的菌株,再作進一步酵素活性的測試及篩選 [1]。
在真核細胞中,也建立了一套細胞表面表現系統。以酵母菌(yeast)為例,其細胞表面表現就優於細菌的系統。許多酵母菌,對人類而言,為GRAS(generally recognized as safe)的有機體,利用其製造出的產品可以用於食物或藥物;其蛋白質分泌(protein secretion)過程,以及轉譯後修飾作用(post-translational modification)也都與高等真核生物相似,非常適合表現真核細胞中複雜的蛋白質,例如:抗體片段(antibody fragments)、細胞素(cytokines)及接受器外表區域(receptor ectodomains);此外,酵母菌具有厚且穩固的細胞壁(cell wall),能夠使的表現於細胞表面的蛋白質維持穩定。到目前為止,酵母菌表面表現系統(yeast surface display system)著重於Saccharomyces cerevisiae的品種,因為對其表現系統、細胞壁結構及細胞壁蛋白質組成有很透徹的研究;而對於其他品種的酵母菌,所能獲得的知識較為缺乏,所以較不拿來應用 [4]。
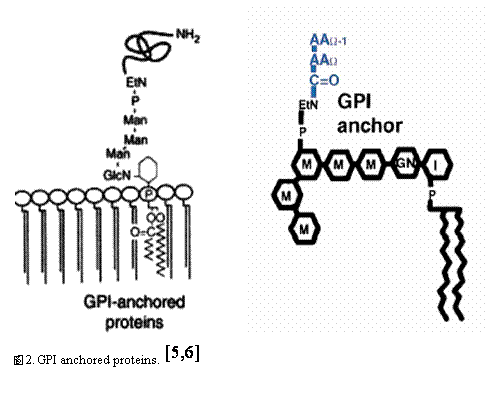
不過隨著研究的累積,最近對於酵母菌中的Hansenula polymorpha的瞭解也逐漸較多。H. polymorpha 為利用甲基的酵母菌 (methylotrophic yeast) ,用其當作細胞表面表現的宿主有下列幾個特性:(1)具有關於甲基代謝(methanol metabolism)很強的啟動子(promoter)。(2)能夠很穩定的將外來的多數基因(multipe copies of foreign genes)維持在染色體中。(3)生長迅速。在Kim等人的研究中,將HpSED1、HpGAS1、HpTIP1及HpCWP1等四種能轉譯出glycosylphosphatidyl-inositol(GPI)-anchored cell surface proteins(圖2)的基因送到H. polymorpha內,希望能建立一套屬於H. polymorpha的細胞表面表現系統。GPI-anchored proteins為真核細胞皆會表現的蛋白質,在S. cerevisiae中已經研究非常透徹,其在細胞表面有很大量的表現,每個細胞表面約有5×104個分子;利用其靠近C端的部分,將其他蛋白質接上,並且送入S. cerevisiae表現都有很好的結果,例如工業上很重要的蛋白質:-amylase、CMCase、-glucosidase及glucoamylase。Kim等人研究結果顯示,HpCWP1所轉譯出的蛋白質-- HpCwp1p具有很高地鑲嵌於細胞表面的能力,可以利用這項研究結果建立一套屬於H. polymorpha的細胞表面表現系統 [4]。
在酵母菌中除了使用GPI--anchored proteins外,還有另外一套系統,就是利用yeast-surface receptor-agglutinin [1]。在S. cerevisiae,-agglutinin是高糖基化的細胞壁鑲嵌蛋白(highly glycosylated cell wall-anchored protein),其表現於交配型(mating type),在交配激素(mating pheromone)-- a-factor的誘導下會有很高的表現;-agglutinin是由AG1所轉譯出來的,650個胺基酸組成的單一polypeptide,其N端具有分泌訊息(secretion signal),C端接上GPI [7]。其也是利用具分泌訊息及鑲嵌於細胞表面的部分,讓重組蛋白表現於細胞表面。
至今,細胞表面表現已經被用來表現peptide、抗體及T細胞接受器(T cell receptor)等,其應用的廣度就跟利用噬菌體表現一樣 [1],作為大量篩選蛋白質的一種方式,使得對於蛋白質的研究能夠更佳迅速。
參考資料
1.H. Lin and V.W. Cornish. Screening and Selection Methods for Large-Scale Analysis of Protein Function. Angew. Chem. Int. Ed. (2002) 41: 4402- 4425.
2.Chen W, Georgiou G. Cell-Surface display of heterologous proteins: From high-throughput screening to environmental applications. Biotechnol Bioeng. (2002) 79(5):496-503.
3.Kramer RA, Brandenburg K, Vandeputte-Rutten L, Werkhoven M, Gros P, Dekker N, Egmond MR. Lipopolysaccharide regions involved in the activation of Escherichia coli outer membrane protease OmpT. Eur J Biochem. (2002) 269(6):1746-52.
4.Kim SY, Sohn JH, Pyun YR, Choi ES. A cell surface display system using novel GPI-anchored proteins in Hansenula polymorpha. Yeast. (2002) 19(13):1153-63.
5.www.hhmi.ucla.edu/C168/week4/lecture2.html
6.Lipke PN, Ovalle R. Cell wall architecture in yeast: new structure and new challenges. J Bacteriol. (1998) 180(15):3735-40.
7.Chen MH, Shen ZM, Bobin S, Kahn PC, Lipke PN. Structure of Saccharomyces cerevisiae alpha-agglutinin. Evidence for a yeast cell wall protein with multiple immunoglobulin-like domains with atypical disulfides. J Biol Chem. (1995) 270(44):26168-77.