蛋白質結構概論
整理:陳威志、卜峰麟
2.蛋白質即是多胜肽鏈(Proteins are polypeptide chains)
3.遺傳密碼對應其特定的20種不同的胺基酸支鏈(The genetic code specifies 20 different amino acid side chains)
4.半胱胺酸能形成雙硫鍵(Cysteines can form disulfide bridges)
5.胜肽單位是構成蛋白質結構的基礎材料(Peptide units are building blocks of protein structures)
6.甘胺酸可以採取許多不同的構形存在(Glycine residues can adopt many different conformations)
7.結論(Conclusion)
8.Motif of protein structure
9.蛋白質的內部為疏水性的(Interior of protein is hydrophobic)
10.α heilx是二級結構的重要元素(The alpha (α) helix is an important element of secondary structure)
11.α helix具有dipole moment(The α helix has a dipole moment)
12.某些胺基酸偏好存在α helix中(Some amino acid are preferred in α helix)
13.組成β sheet的β strands可以是同向排列或是反向排列(Beta(β)sheets usually have their beta strands either parallel or antiparallel)
14.環狀區域會位在蛋白質分子的表面(Loop regions are at the surface of protein molecules)
重組DNA的技術可以當作一種工具去很快的定出DNA序列,且藉由構造基因來推出蛋白質的胺基酸序列。現在已經有愈來愈多的序列被定義出來,但是卻很少知道這些序列在生物系統上的功能。我們在自然界中發現的蛋白質是演化的產物;透過選擇的壓力,而演化出執行特定功能的蛋白質;然而,這些功能又與它們的三維結構有關。這些三維結構是由多胜肽鏈中的胺基酸序列摺疊產生的結果,從直鏈形的構造摺疊成緊密的domains,而這些domains有著特定的三維結構(圖1)。摺疊成的domains可以當做組件而組成較大的assemblies,如病毒或肌肉纖維;除此之外,還可以當作酵素的特異催化位置,或當作某些用來攜帶氧氣或調節DNA功能之蛋白質的結合位置。
為了了解蛋白質的生物功能,人們可以從胺基酸序列所構成的三級結構中去預測或推論出來。但在過去二十五年中,有關摺疊的問題還一直不為人們所了解,這對於分子生物學家是一個相當大的挑戰。因為每個蛋白的三級結構無法被直接觀察到,因此人們利用x射線晶體學(x-ray crystallography)或核磁共振儀(NMR)的技術來測知,在過去三十年,有將近500種蛋白質已經利用x-ray的方法測定出來了。當人們研究的愈透徹,就可以愈清楚地知道蛋白質的構造如何產生、與構造相關的作用為何,並可以知道不同蛋白質之間的一些基本關聯等。
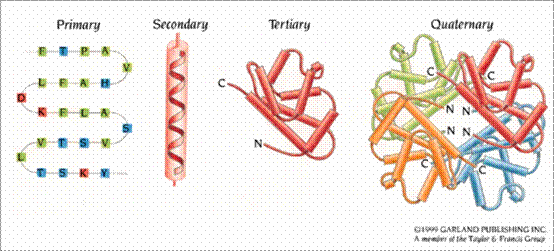
圖1: 蛋白質的胺基酸序列稱為一級結構,而序列的不同區域則形成局部規則的二級結構,例如αhelix 或β strand。三級結構則是由此類結構要素,包裹成一個或多個緊密的球狀單位所形成,這些單位稱為domains。最終的蛋白質可能包含數個多胜肽鏈排列在四級結構中。藉著形成此類三級和四級結構,使原本位在序列上距離很遠的氨基酸,在三度空間中靠攏,以形成有功能的區域(active site)(1)。
2.蛋白質即是多胜肽鏈(Proteins are polypeptide chains)
20個胺基酸全部都有一個中心碳原子(Cα),其上有氫原子(H)、胺基(NH2)以及羧基(COOH)與之鍵結。而胺基酸之間的不同,在於Cα第四個位置所接的支鏈(side chain) (圖2)。20種不同的支鏈(side chains)是由特定的遺傳密碼(genetic code)所轉錄轉譯而來,但有些很少數的情況是於轉譯後,再經酵素修飾的產物。
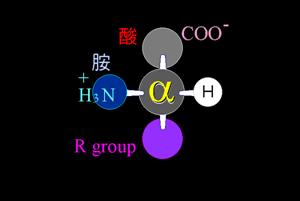
圖2 : 胺基酸的基本構造(6)。
胺基酸在蛋白質合成的時候,藉由胜肽鍵(peptide bond)與其他胺基酸做連結。一個胺基酸的羧基(carboxy group),會與下一個胺基酸的胺基(amino group),行脫水縮合,形成一胜肽鍵;這個過程會一直重複,以使多胜肽鏈不斷的延伸。最後,多胜肽鏈(polypeptide chain)之第一個胺基酸的胺基和最後一個胺基酸的羧基都會被原封不動地保留著。而形成的連續peptide bond則構成主鏈(main chain),或稱為骨幹(backbone),再由此伸出各種支鏈(side chain)。
主鏈的Cα原子上,會接有一個NH group、一個羰基(carbonyl group,C’=O),和一個氫原子;其中,羰基是以C’與Cα做鍵結。這樣的單位(或稱residues)會藉由胜肽鍵(peptide bond)的連接,而形成一條多胜肽(polypeptide);而胜肽鍵指的是,一個單位的C’原子與下一個單位的氮(nitrogen)原子之間產生的鍵結。主鏈最基本的重複單位,不論從生化或基因觀點來看,就是(NH - CαH - C’O);此亦為胺基酸在形成peptide bond後的共同部分。
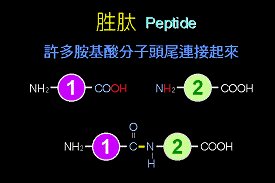
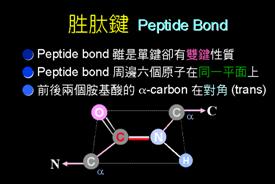
圖3 : 胜肽(Peptide)是由許多胺基酸分子所連接而成。由前一個胺基酸 (1) 的羧基與後一個胺基酸 (2) 的胺基,經脫水反應形成鍵結;此產生的新鍵,稱為胜肽鍵 (peptide bond)。而所生成的雙胜肽 (1-2) 還有一個胺基及羧基,可以繼續連接下去(6)。
3.遺傳密碼對應其特定的20種不同的胺基酸支鏈(The genetic code specifies 20 different amino acid side chains)
有20種不同的胺基酸支鏈會出現在蛋白質裡,它們的名字可縮寫成三個字母或一個字母的代號。根據支鏈的化學特性不同,胺基酸通常分為三種類型,第一類是帶有疏水性的支鏈(hydrophobic side chain),如Ala (A)、Val (V)、Leu (L)、Ile (I)、Phe (F)、Pro (P)、Met (M);第二類是帶有電荷的Asp (D)、Glu (E)、Lys(K)、Arg ( R);而第三類是帶有極性(polar)支鏈的Thr (T)、Cys (C)、Asn (N)、Gln (Q)、His (H)、Tyr (Y)、Trp (W)。胺基酸glycine (G)的支鏈只有一個氫原子,因此也是20種胺基酸裡面構造最簡單的一個。它具有特殊的性質,通常被當作第四類的胺基酸,或者是歸類到第一類中。
除了glycine有兩個氫原子與Cα鍵結外,所有胺基酸的Cα上接的四個原子團都是不同的。因此,除了glycine外,所有胺基酸都是對掌性分子(chiral molecule),而有L form與D form之分。
生物系統依據分子特異性來做辨別,其中包含chiral form的不同。用於蛋白質合成的轉譯系統,在演化的結果下,只選用了胺基酸的其中一種chiral form---L-form。然而,為什麼在演化的過程中,被選擇的是L-form,而不是D-form,至今也尚無明確的答案。
4.半胱胺酸能形成雙硫鍵(Cysteines can form disulfide bridges)
位在多胜肽鏈(polypeptide chain)中不同部分的兩個cysteine residues,若兩者在蛋白質的三維結構中,位置很靠近,則可以被氧化而形成雙硫鍵。此反應需要在氧化的環境中進行,因此細胞內的蛋白質(intracellular proteins)通常不會有此類的雙硫鍵產生,因為細胞內的蛋白質所處的環境是一還原的狀態。然而,雙硫鍵卻常發生在細胞外的蛋白質(extracellular proteins)中。在真核細胞中,這些雙硫鍵的形成,發生在內質網的腔(endoplasmic reticulum lumen)內,此處為分泌路線(secretory pathway)的第一個位置。
雙硫鍵可以使三維結構穩定。在一些蛋白質中,這些鍵結可以使不同的多胜肽鏈(polypeptide chain)結合在一起;舉例來說,insulin的A chain和B chain即是藉由兩個雙硫鍵來做連結的。通常,在分子內的雙硫鍵可穩定一條多胜肽鏈摺疊後的構造,以使蛋白質不易被分解。除此之外還有很多的例子,例如蛇毒液中的毒素和protease inhibitors。
5.胜肽單位是構成蛋白質結構的基礎材料(Peptide units are building blocks of protein structures)
前文中有提到一種方式,能將多胜肽鏈的主鏈,區分為許多重複的單位;而此處又以另一種方式加以區分,目的是為了使人們更容易去描述蛋白質的結構特性。此處的一個胜肽單位(peptide unit),乃是指從一個Cα到下一個Cα。因此,除了第一個和最後一個Cα原子外,每一個Cα原子都同時屬於兩個這樣的單位。之所以要這樣劃分,乃是因為每個小單位裡面,所有的原子都可以固定於同一個平面上;而且,所有蛋白質中的每個單位裡的鍵長和鍵角都幾乎相同。但須注意的是主鏈的胜肽單位中,並沒有包含不同的支鏈結構。作者將選擇不同的方式來解釋多胜肽鏈的生化與構造特性,並討論不同胺基酸序列所構成的蛋白質,和planar peptide units的序列。
胜肽單位是由一群堅硬的原子團所組成,彼此間靠著共價鍵,連接成一條多胜肽鏈。它們僅有的自由度(degree of freedom)即位於此兩個共價鍵鍵結的位置(Cα- C’和N - Cα)。每個單位可以繞著此兩個鍵的地方做旋轉(圖4)。對於一Cα原子,在N - Cα鍵所構成的旋轉角度稱為phi(ψ)角,而在Cα – C’所構成的角度稱為psi(Ψ)角。如此amino acid residue即是由此兩個構造角度連結起來的,這是唯一可以自由變換的地方。當每個胺基酸的ψ角和Ψ角決定後,整個polypeptide主鏈的結構也將被決定。
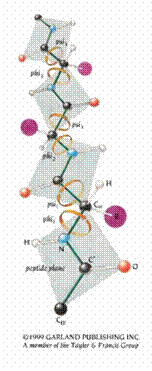
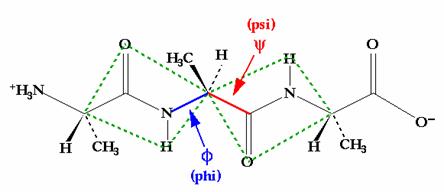
圖4 : 圖中多胜肽鏈的主鏈之原子是以堅硬的胜肽單位(peptide units)描繪之,而每個胜肽單位間則是藉由Cα原子加以連接。每一單位具有兩個自由度(degree of freedom),即它能繞著兩個鍵結做旋轉,分別是Cα-C’鍵和N-Cα鍵。繞N-Cα鍵旋轉的角度稱為phi(ψ),而繞Cα-C’鍵旋轉的角度則稱為psi(Ψ)。因此,主鏈原子的形態決定於每一胺基酸的這兩個角度的數值(1)。
圖5 : 由alanine構成的tripeptide。中心Cα原子兩側的peptide bond可使兩邊堅硬的平面(rigid plate)能分別以phi(ψ)和psi(Ψ)做旋轉(4)。
6.甘胺酸可以採取許多不同的構形存在(Glycine residues can adopt many different conformations)
在胺基酸中,大多數ψ角和Ψ角的組合並不被允許,這是因為支鏈和主鏈間的空間障礙(steric collision)。因此,很容易就能計算出能被允許的組合有哪些。因為胺基酸的D-form和L-form裡,其COO- group的向位不同,因此它們也會存在不同的ψ角和Ψ角。如果D amino acid存在,則它們所形成的蛋白質結構,會與現在於自然界中發現,由L amino acid所構成的蛋白質的結構不同。
印度生物物理學家G. N. Ramachandran,是第一個計算出sterically allowed region的人。他還以ψ角和Ψ角為座標軸,創作出Ramachandran plot(註)。它可以精確地從蛋白質的結構裡畫出除了glycine外的所有胺基酸。因為Glycine的支鏈是由氫原子所組成,所以它可以較其他的residues組成更多種的構型。Glycine在造構上扮演一個很重要的角色,因為它可以允許一些不常見的主鏈構型(unusual main chain conformation)存在於蛋白質中;這也是為什麼在homologous protein sequences中,會有很高比例的glycine residues存在。
7.結論(Conclusion)
所有蛋白質分子是由二十種不同胺基酸,藉由胜肽鍵(peptide bond)所連結成的聚合物。構造基因的核苷酸序列決定胺基酸序列,而胺基酸序列決定蛋白質的三維結構,最後蛋白質的結構則決定了它的功能。每個胺基酸中,都由一些相同的原子來組成主鏈的部分,然後再由剩下的原子來構成具有厭水性、極性或帶電的支鏈。整個蛋白質的主鏈結構,是由每個胺基酸中的兩個結構角ψ角和Ψ角所決定。對於這些角度,通常只有特定的組合才可以存在,這是因為在主鏈和支鏈之間會有空間障礙的存在。
G. N. Ramachandran利用polypeptide的電腦模組改變ψ角和Ψ角的組合,最後找尋穩定的構形組態。對於每種的構形,必須檢查原子與原子間的聯結狀態,而每個原子當作是一個堅硬的球體,而直徑則相當於它們凡得瓦力半徑。因此ψ角和Ψ角會使球體相互抵觸,直到找到polypeptide的sterically allowed conformation。
Ranmachandran plots展現出結構中所有residues的ψ角和Ψ角之扭轉角度(不包含chain termini)。在圖中所代表的區域,黃色區域內黑點存在的地方是表示ψ角和Ψ角最容易的組合方式,而藍色區域外面則表示sterically disallowed conformation的區域。理想的情況下,希望有90%以上的residues可以位於圖上的黃色核心區域。而residues落在核心區域裡的百分比,則可作為stereochemical quality的一個指標。
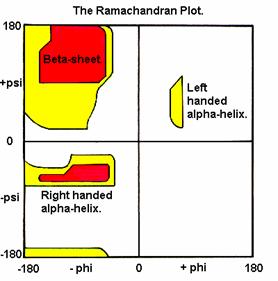
圖6
: The Ramachandran plot(2)。
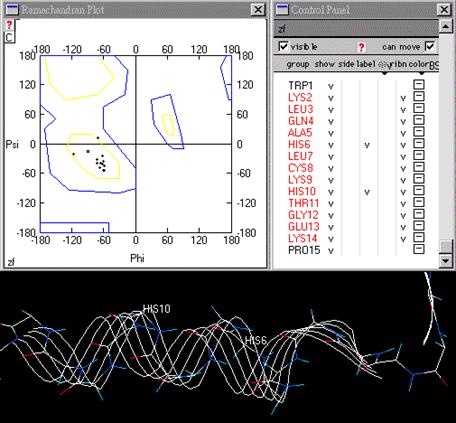
圖7 : Alpha helix的Ranmachandran plot分析圖。
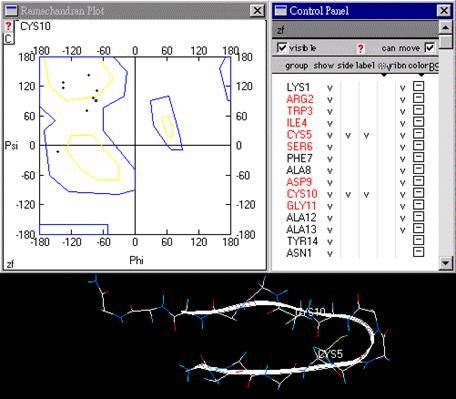
圖8 : Beta sheet的Ranmachandran plot分析圖。
8.Motif of protein structure
X-ray結構的研究,可以用來預測未知分子的特性。當W. L. Bragg解出NaCl的晶體結構後,改變了當時人們對於離子化合物的鍵結力的觀念。第一個利用x-ray繞射的方法解出的蛋白質晶體,為球狀蛋白---myoglubin,此一發現使得原本以為掌握蛋白質折疊與其功能的研究者,感到震驚。在過去的研究中,我們可以輕易的了解蛋白質之所以能夠執行其多樣性的功能,結構上的不規則性佔了很大的因素,不像儲存於DNA的訊息與其所轉譯出的訊息,是成線性的關係。因此DNA的結構可以很相似,卻帶著不同的遺傳訊息,也就是說DNA的結構與其所帶的資訊是獨立的;至於蛋白質用來辨認細胞內成千上萬不同種類的分子,是藉由需要多樣和不規則的三度空間結構所造成的細部交互作用。雖然有多樣性以及不規則的結構,但還是有規則性的存在,例如:二級結構。而解出二級結構的學者也提出了一個觀念:「一個分子最值得注意的特色,或許在於其複雜性以及缺乏對稱性;且其複雜程度超過任何一種蛋白質結構理論所預測出來的結構。」
9.蛋白質的內部為疏水性的(Interior of protein is hydrophobic)
對myoglobin作更深入的研究後,發現其蛋白質內部幾乎全都是帶有厭水性side chain的胺基酸。而摺疊水溶性球蛋白最主要的驅動力,是將疏水性的side chain包裹到分子的內部裡,產生一疏水性的核心(core)及親水性的表面(surface)。然而,一protein chain在產生此類疏水性核心的過程中,會遇到一個問題,也就是:為了將厭水性的side chain摺疊進蛋白質內部,主鏈也必須同時被摺疊進去。而主鏈因為每個peptide unit,都含有一個hydrogen bond donor(NH)和一個hydrogen bond acceptor(C’O),所以極性很高,也就是親水性很高。在疏水性的環境中,這些主鏈上的極性groups,必須藉著形成氫鍵來中和其極性。此問題即藉著在蛋白質分子的內部形成規則的二級結構而得到巧妙的解決。此類二級結構通常是α helix或β sheet兩種形式的其中一種。而此兩種不同的二級結構都是藉由主鏈上的NH和C’=O group互相形成氫鍵而形成的。必須當許多連續的residues具有相同的phi(ψ)、psi(Ψ)angle時,這些二級結構才會形成(圖4)。以下就讓我們更進一步的探討這些重要的結構組成。
10.α heilx是二級結構的重要元素(The alpha (α) helix is an important element of secondary structure)
α helix為蛋白質結構的典型組成,這是1951年Linus Pauling在California Institute of Technology第一次提出的。他預測α helix在蛋白質中會是個很穩定、很適合的結構。此預測很快地就由Max Perutz從hemoglobin的晶體以及keratin纖維的繞射圖形(diffraction pattern)中獲得了強有力的實驗支持。而John Kendrew對myoglobin結構的高解析研究,則給了此預測更徹底的證明;他發現myoglobin中所有的二級結構都是螺旋形的(helical)。
當一串連續residues的phi(ψ)、psi(Ψ)angle都接近於-60度和-50度時(即相當於Ramachandran plot中,左下方的象限),就可以形成α helix的結構。α helix每一圈含有3.6個胺基酸,且於n residue的C’O group和(n+4) residue的NH group之間會形成氫鍵。因此,除了第一個NH 和最後一個C’O group外,所有的NH 和C’O group都會參與氫鍵的形成。而結果使得α helix的末端具有極性,所以大多會位在蛋白質分子的表面。
α helix在球蛋白中的長度變化可以很大,從4、5個胺基酸到超過40個residues都有。平均長度約為10個residues,也相當於三個turn。α helix中,每升高一個residue就是1.5 Å,所以一般平均的α helix約15 Å長。α helix理論上可以是右旋(right-handed)或是左旋(left-handed)的,但是實際上在蛋白質中觀察到的α helix幾乎都是右旋的形式,這是因為L-form胺基酸其side chain和C’O group太過靠近而無法形成左旋的α helix。而3-5個residues長度的左旋α helix會偶爾發生。
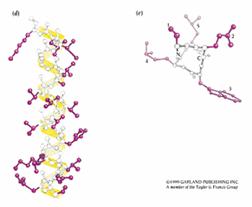
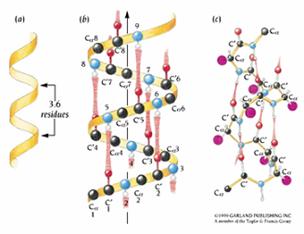
圖9 : α helix為一種主要的蛋白質二級結構。 (a) α helix的理想圖。每一個turn有3.6個residues。 (b)和(a)相同,並標示了主鏈的原子,以及氫鍵的位置。 (c) αhelix的分子模型結構。(d) myoglobin中一個α helix的stick-and-ball model。紫色的部份為side chains。 (e) αhelix的一個turn,紫色代表side chain的位置(1)。
11.α helix具有dipole moment(The α helix has a dipole moment)
α helix中的氫鍵,都為同一個方向,所以形成的peptide unit也為同一個方向,皆與helical axis的方向相同。而peptide unit具有偶極矩(dipole moment),是因為NH和C’O group不同的極性大小所致。同樣的,這些偶極矩也是沿著螺旋的中心軸線排列。此效應最後使得α helix的amino end呈現partial positive charge,而carboxy end則呈partial negative charge。這些電荷可以吸引帶相反電荷的ligand。帶負電的ligands(尤其是帶有phosphate group的ligand),常會結合到α helix的N terminal;但是,帶正電的ligands卻很少會結合到α helix的C terminal。可能的原因是:除了N terminal的dipole effect外,其free的NH groups在幾何學上也具有很適合phosphate group結合的位置。
12.某些胺基酸偏好存在α helix中(Some amino acid are preferred in α helix)
α helix結構中,胺基酸的side chain都會向外突出(圖9 e),因此不會破壞αhelix的結構,但proline例外。因為proline為環狀結構,其side chain的最後一個原子與主鏈上的N原子形成鍵結,使得N原子無法參與氫鍵的形成,並且造成了α helical conformation空間上的障礙。proline可以在α helix的第一個turn中很穩定的存在,但在任何其他位置時,通常都會使α helix產生很顯著的彎曲(bend)。這樣的彎曲存在於許多α helix中,但不一定都含有proline在其中。因此,雖然我們可以預測proline residue可能會造成α helix的彎曲,但並不是所有的彎曲都是proline所造成的。不同的胺基酸side chain中,也些偏好形成α helix有些則不然。其中,偏好形成α helix的胺基酸有:Ala、Glu、Leu和Met;而Pro、Gly、Tyr、和Ser則非常不喜歡形成α helix。α helix通常位於蛋白質的外圍,帶有極性的一側面向水溶液,而另一側則面向厭水性的蛋白質內層。由於每一圈有3.6個residues,因此每3~4個residues就會由厭水性的side chain轉為親水性的。這樣的趨勢有時候能在胺基酸序列中看到,但是光用它無法當作可信賴的結構預測法,因為面向水溶液的residues也可以是疏水性的,而且α helix也可以完全埋藏在蛋白質中或完全暴露出來。表一中顯示的例子,分別是代表完全埋藏、部分埋藏和完全暴露型的α helix之胺基酸序列(表一)。
Helical wheel or spiral是一個很方便的圖示法,可以用來說明helix中的胺基酸序列。因為α helix一個turn有3.6個residues,所以每隔100度(360°/3.6 = 100°)就可以畫出一個胺基酸(圖11)。

表一: 三個α helix的胺基酸序列
第一個序列是從citrate synthase(260-270 residues)來的,為一buried helix。第二個序列來自alcohol dehydrogenase(355-365 residues),為partially exposed helix。第三個序列來自troponin C(87-97 residues),為completely exposed helix。紅色表帶電的residue,藍色表極性residue,綠色表疏水性residue。
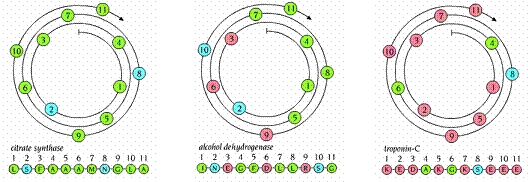
圖11 : 表一中peptide chain的胺基酸序列以helical wheel表示之。沿著螺旋,每隔100度畫一個胺基酸。顏色表胺基酸side chain的特性,綠色表疏水性,藍色表極性,而紅色表帶有電荷。
13.組成β sheet的β strands可以是同向排列或是反向排列(Beta(β)sheets usually have their beta strands either parallel or antiparallel)
第二種主要的二級結構為β sheet,此結構是由peptide chain中的數個區域相互結合而成;而α helix則是由一連續的區域所形成的。β strand通常為五到十個胺基酸長,位於Ramachandran plot中的左上方象限,且structural allowed region佔的範圍很廣(圖6)。β strand會相互毗鄰而排,因此一strand的C’O group可與鄰近strand的NH group相互形成氫鍵。而數條β strand結合在一起,就形成了β sheet;又因為Cα原子會鏈續在β sheet平面的上下變化,所以β sheet會呈現一摺板狀(pleated)。其side chain也會跟隨此pattern,而位於β strand的上方或下方。β sheet分成兩種,若β strand間相連接的方式為同一方向,均由N-terminal到C-terminal,則為parallel;若為不同的方向,則稱為antiparallel。這兩種形式的β sheet,其氫鍵形成的pattern也不同。Antiparallel β sheet其氫鍵間形成的空間會有大小的變化(圖12);而parallel β sheet所形成的空間則是均等的(圖13)。有些β sheet也可以由antiparallel和parallel兩種形式所組成,但機會較少。圖14則說明mixed β sheet之氫鍵在β strand中是如何排列的(圖14)。幾乎所有形式的β sheet,其β strand均有扭轉的現象,而扭轉的方向皆會相同。
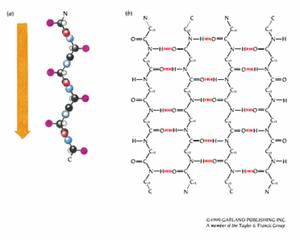
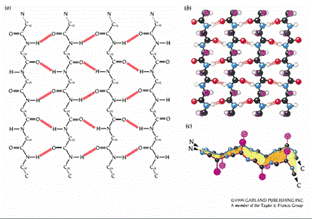

圖14 : Mixed βsheet。此蛋白為大腸桿菌的thioredoxin。(a) twist of βsheets。 (b) mixed β sheet 中,β strands之間的氫鍵位置。
14.環狀區域會位在蛋白質分子的表面(Loop regions are at the surface of protein molecules)
大部份蛋白質的結構均是由α helix和β strand 所組合而成,兩者之間靠著不同長度和形狀的loop regions連接起來。二級結構的組合在分子內部形成穩定的厭水性中心,而loop regions則位在分子的表層。loop regions之主鏈上的C’O和NH groups原則上不會形成氫鍵,而是暴露在溶劑中與水分子形成氫鍵。而暴露於溶劑中的loop regions多為帶電性或是極性(親水性)的胺基酸。可以利用此一特性,從胺基酸的序列來推斷是否為loop region,這樣的預測,準確性會比預測αhelix和β sheet的準確度要來得高。來自不同種(species)的同源 (homologous) 胺基酸序列,只有在loop regions的地方會發生insertion或是deletion。而蛋白質的中心部份(core)在演化的過程中,發生改變的機率會比loop region來得小。loop region除了連接兩個二級結構外還可以形成binding site或是酵素的活性區(active site)。例如:抗體要和抗原結合的位置就是由六個loop region所組成的,而不同的抗體其loop region的長度和胺基酸序列也都會不同。若loop region連接的是兩個不同方向(antiparallel)的β strand,則此loop-region就稱為hairpin loops,而抗體上和抗原結合的位置就為此種loops。
15.參考資料
1. Branden, C., Tooze, J. 1991. Introduction to protein structure, p.3-p.19, 1st ed. Garland Publishing, Inc., New York.
2. http://www.cryst.bbk.ac.uk/PPS95/course/3_geometry/rama.html
3. http://www.biochem.ucl.ac.uk/~roman/procheck/manual/examples/plot_01.html
4. http://www.cgl.ucsf.edu/home/glasfeld/tutorial/AAA/AAA.html
上述的三個網址都是在介紹Ramachandran plot,有原理介紹也有背景等詳細的資訊,其主要歸屬於蛋白質幾何學的網頁中,他主要的首頁是VSNS-PPS(The Principles of Protein Structure)由Birkbeck College和Virtual School of Natural Sciences(VSNS) of the Globewide Network Academy (GNA)所共同研究建立的網站。
5. http://binfo.ym.edu.tw/post/intro/ramachan_plot.htm
6. http://140.112.78.220/~juang/BCbasics/index.htm
上述的兩個網址是台大農化系莊榮輝老師的網站,有非常詳細的細胞、生化相關資料。