蛋白質體學的研究方法
整理:施純如
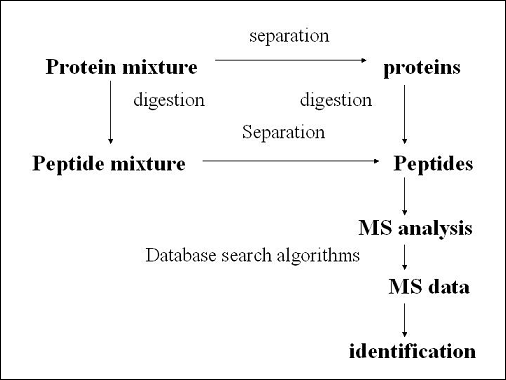
要將蛋白質樣品送入質譜儀(MS)分析,首先要做兩件事,第一,必須將蛋白質分解為較小單位的peptides,這個步驟可以使用蛋白水解酶達成;第二,要將原本非常複雜的蛋白質(或peptides)分離成較不複雜的混合物。在這兒,我們可以選擇:先分離純化混合的蛋白質再利用酵素將其水解成較小peptides,之後分析peptides產物;或是先水解原本複雜的蛋白質混合物,再將peptides分離,並分析分離過後的peptides。至於要選用哪種方法,可以視情況而定,各有利弊。
實際的研究中,我們的樣品通常是一塊組織的切片、一盤培養的細胞、一管細菌培養液,或是一片葉子。在蛋白質體的研究中,由生物檢體、細胞或細菌培養液萃取出蛋白質的方法有許多種,共同的步驟是弄碎樣品、再用超音波振盪等方法使其均質化,並盡量去除這些組織液、細胞液或菌液的雜質以及其它汙染;由於研究的對象是蛋白質,故要去除的包括脂質、纖維素和核酸……等,這個目的可由加入以下的試劑達成:
1. 界面活性劑(detergents):像SDS, 3- ([ 3-cholamidopropyl]dimethylammonio)-1-propane sulfonate ) (CHAPS), cholate, Tween),可以協助將蛋白質與脂質分開,利於膜蛋白的分離。
2. 還原劑(reductants):如dithiothreitol [DTT], mercaptoethanol, thiourea,可以防止蛋白質氧化並破壞雙硫鍵。
3. 變性劑(denaturing agents):如尿素和酸性物質,運用離子強度和極端的pH值,阻礙蛋白質二級與三級結構的產生,可以防止蛋白質之間的交互作用(protein-protein interaction)。
4. 酵素:如DNAse、RNAse,用以去除核酸、醣類或脂質。
目前學者們已經研究出由不同樣品中,萃取出蛋白質的方法,有些時候除了上述藥品外,也加入一些蛋白酶的抑制劑,以防止蛋白質在萃取過程被水解。值得注意的是,無論是界面活性劑、還原劑、變性劑,或是蛋白酶抑制劑,不能排除它們對蛋白質體結果分析的影響,如phenylmethylsulfonyl fluoride(PMSF)這種serine protease 的抑制劑,它會影響後續的胰蛋白酶分解作用,界面活性劑也會對蛋白質的分離有負面效果。因此,收集樣品的資料十分重要,必須確定這些試劑的使用,不會對最終的分析造成太大的影響。
要將細胞組織萃取出的數千種蛋白質分開,有三種主要的方式:1D-和2D-SDS-PAGE以及IEF(isoelectric focusing),另一種選擇是使用高效能液相層析(HPLC)。二維電泳,也就是2D-SDS-PAGE,所依據的原理是各蛋白質等電點和分子量的不同,同時進行isoelectric focusing(IEF)和SDS polyacrylamide gel electrophoresis,二維電泳完成後,可以將蛋白質保存於膠體中,直到下一個分析步驟。
跑完膠體電泳後的工作,分為以下幾項:
l 比較treated和non-treated樣品在2-Dgel 上的差異
l 偵測新的、消失的,或是改變表現量的蛋白質
l 將蛋白質點量化
l 對準有差異的蛋白質點切下分離
實驗組與對照組蛋白質樣品在膠體上跑出來的差異性,要以肉眼精確分辨出來是不可能的,因此,膠體的影像必須藉由數位相機或掃描器轉為電腦可辨識的檔案(通常是灰階的TIFF檔),使用軟體分析色帶的深淺差異,也就是蛋白質表現量的多寡。

左圖為 ImageScanner II,適用於Coomassie 或silver 染法的膠體掃描器
右圖為Typhoon,適用於螢光、冷光、放射線標定染法的膠體掃描器
掃進電腦後,使用ImageMaster software 這套軟體可以偵測膠體上面各個蛋白質點、去除背景值,以及比較不同膠體相同位置上蛋白質點的多寡,將這些資訊加以儲存。
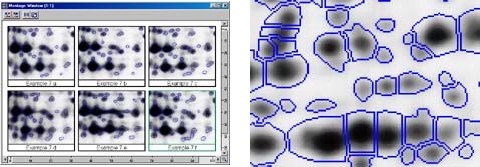
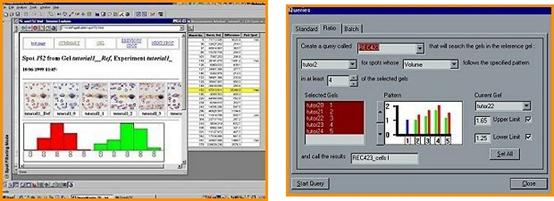
之後,可以選擇目標蛋白質點,以spot picker挑出, 機器會將它轉移到培養盤,方便之後酵素切割等實驗操作。使用ImageMaster 2D Elite Software 可以藉由機器判斷或人工挑選的方式,列出「protein pick list」,這份清單傳送到spot picker後,即可正確挑出上面所列的蛋白質點。Spot picker一般可以放置280x250mm大小、1~1.5mm厚度的膠體,picker尖端沾取膠體的部份,也提供不同尺寸供選擇,可視情況選購。

Ettan Spot Picker
樣品送入質譜儀之前,要先用酵素將大分子的蛋白質切成小片段,這樣有利於儀器作出正確的判斷,因為由6到20個氨基酸所組成的peptides對於質譜儀和database的解讀最為有利也較精確。少於6個氨基酸所組成的序列,就很難在database搜尋中找出相符合比對的結果,多於20個氨基酸的peptides,則是在質譜儀很難獲得序列的正確資訊。
自然界存在上千種已純化出來的蛋白酶,但大部份都只能作少量的純化。用於蛋白質體研究切割的酶,必須是能在大量純化的同時,又能獲得極純的產物,並且切點明確具有專一性、無旁支反應(side reaction)、性質穩定及高效率。下表是符合以上條件、目前常用的蛋白酶:
|
酵素名稱 |
切割點 |
|
Trypsin |
/K-, /R-, \P |
|
Chymotrypsin |
/W-, /Y=, /F-, \P |
|
Glu C (V8 protease) |
/E-, /D-(註), \P |
|
Lys C |
/K-, \P |
|
Asp N |
/D- |
註: 在磷酸鈉(sodium phosphate)緩衝液中,切於aspartate 和 glutamate之後,其它溶液中只切於glutamate之後。
Trypsin
在蛋白質體學的分析中,trypsin是用得最多的蛋白酶,它是一種水解serine氨基酸的酵素,主要來源是牛的胰臟,優點是易於純化得到較多的產量。為了抑制它隱藏的chymotrypsin活性,一般可以用tosylphenylalanylchloromethane(TCPK)加以修飾。Trypsin的切點在蛋白質lysine和arginine的部份,不過,如果這兩個部份在C-terminal方向連接的是proline,它就不會水解這個地方的鍵結。由於許多蛋白質中富含lysine和arginine這兩種氨基酸,所以typsine作用之後得到的片段,很適合用於質譜儀分析,一般而言,50kDa的蛋白質用trypsin切了之後,約可以產生30個peptides。無論是在溶液中,或是在電泳膠體上,甚至是轉印(transfer)後的硝化纖維膜上,trypsin的作用活性都很好,這也是它被廣泛使用的原因。
Glu C (V8 protease)
在ammonium bicarbonate或ammonium acetate 的緩衝液中,Glu C可以切glutamate的carboxyl端,在phosphate類的緩衝液中,它切點變成glutamate和aspartate。這種酵素主要用於膠體中,和trypsin比起來,Glu C的切點特異性較高,可以保持peptide片段的完整性,如果一蛋白質的某部份lysine和arginine含量很高,使用trypsin會導致切得的片段過短,此時就可以考慮用Glu C。
其它的蛋白酶
Chymotrypsin、Lys C、Asp N以及其它一些切點非特異性(nonspecific)的蛋白酶(Subtilysin、pepsin、proteinase K和pronas)偶爾也用於蛋白質體學研究中。其中有些只切少數、甚至只有一種氨基酸,切點很少,因此酵素作用完,得到的peptide片段較大,往往不適用於質譜儀分析;相反的,chymotrypsin因為同時會切tyrosine、phenylalanine和tryptophan這些氨基酸,切點過多,產生的peptides太短,也不適合分析。所以這些蛋白酶,只在特殊情況下使用,例如某個我們有興趣的蛋白質片段,本身不帶有trypsine的切點,便能使用這些較不常用的酵素。
Cyanogen Bromid
蛋白質也能用一些化學物質達成分解的效果,cyanogen bromide(CNBr)便是其中一例,它的切點位於蛋白質methionine的部份,因為只辨識這種氨基酸,作用完的片段往往過於龐大,不適用於質譜儀分析。
將我們有興趣的蛋白質點從膠體上切出,去染色後再加入酵素使其分解成peptides,最後再將peptides從膠體中洗出,而加入酵素使蛋白質分解成小的peptides的過程及稱為「digestion」。
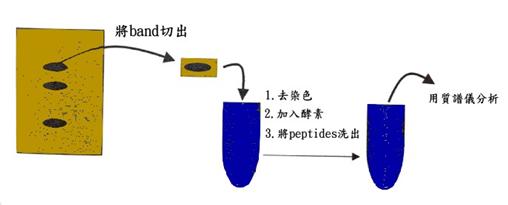
要注意的是,膠體的染色劑若是酸性(如acetic acid),易使蛋白質固定在膠體上,不易被酵素分解,分解出來的peptides也較不容易從膠體分離;膠體硬度高、成份連結緻密者,酵素也不容易滲入和蛋白質作用;另外,膠體的成份中,SDS和未形成複合體的acrylamide(unpolymerized acrylamide),都會影響蛋白酶的活性。
將蛋白質混合物以2-D電泳分開後,惟有用影像分析方式,才能知道蛋白質是否產生表現量的變化,但是這樣的分析並不足以讓人鑑定出蛋白質的身分,要辦別蛋白質的身分,必需使用質譜儀(mass spectrometry),分析膠體上蛋白質形成的點,分析完成後的資料再去比對已有的資料庫才能正確的找到蛋白質的身分。
目前有兩種設備用於蛋白質體學的質譜儀(MS)實驗,一種是MALDI-TOF MS(matrix-assisted laser desorption inoization-time of flight mass spectrometry),一種是ESI-tandem MS(electrospray ionization-tandem mass spectrometry),前者可獲得peptide質量的資訊,後者可獲得peptide片段詳細的資料。雖然兩種操作的方式截然不同,但得到的結果都具有高度準確性,專門研究蛋白質體學的實驗室,會將兩種皆列為必要的設備。
MS本身有三個主要部份,如下圖所示:
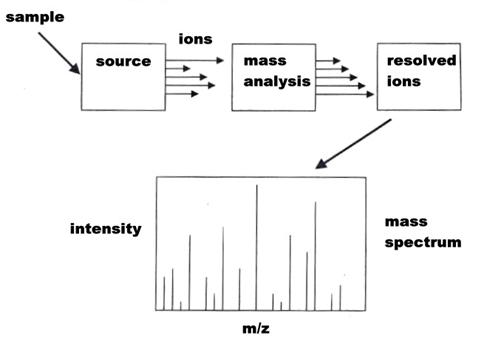
第一個部份是來源(source),由樣品產生離子;第二個部份是質析(mass analyzer),依離子質量/價數【mass/charge(m/z)】的比例分開它們;第三個部份是偵測(detector),也就是mass analyzer的結果。簡單說來,質譜儀就是將一群混合物轉為離子,再依照m/z分析它們,得到的結果由儀器自動記錄下來,交給電腦分析。
怎樣的MS適合?有三個重點,第一是敏感度(sensitivity),多數的蛋白質體研究中,所能得到的蛋白質量都有限,所以儀器最好能敏感到能偵測10-15莫耳的peptide;第二是解析能力(resolution),從極小的m/z值分辨不同離子,最好能區別m/z值差別在0.001 amu以下的樣品,限於經費,這樣的儀器其實不普遍,常用的大概在1 Da(一個氫原子的質量)左右;第三是精密度(mass accuracy),測得的peptide離子或片段愈接近真實狀況愈好。
五、Database的搜尋
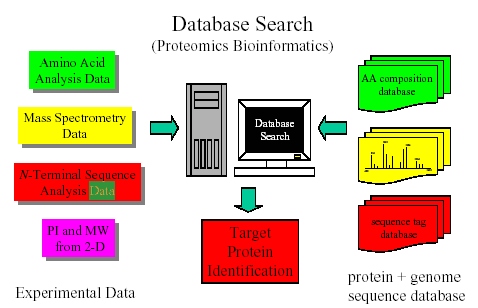
將蛋白酶切割後的產物送入質譜儀,可測出peptide的質量(mass),接著利用生物資訊工具,以網路連到蛋白質資料庫,可以查詢用什麼酵素水解哪一種蛋白質後會得到那一些peptide片段,比對由質譜儀得到的結果,看看有沒有符合的胺基酸序列,即可知道treated與non-treated 樣品,會改表什麼蛋白質的表現量。如果在peptide和protein database沒有搜尋到相符合的,可以查詢genomic database,DNA 的open reading frame在理論上是可以轉為胺基酸序列的。透過質譜儀分析,除了可以得知氨基酸序列,也可以獲得簡單的蛋白質結構,像是雙硫鍵的存在、轉錄後的磷酸化、乙醯化或醣化修飾。知道待測蛋白質的組成和一些結構的資訊後,便能推測它可能具有的功能。
Daniel C. Liebler Introduction to Proteomics-Tools for the New Bioloogy p31~56
Humana Press Inc. (2002)